От начала до конца
Задача
Проснувшись, один юный биоинформатик с нетерпением дожидался темноты, поскольку днем ему редко что-нибудь удается. Лишь с последними лучами солнца он открыл крышку ноутбука и приступил к работе: скрипт, второй... Всё начинается с решения простейших житейских задач для разминки, и на повестке дня вопрос: точно ли все гены начинаются старт-кодоном AUG, как пишут об этом в учебниках, и заканчиваются одним из трех стоп-кодонов? Глупо, конечно, но этим вечером было решено начать именно с такой задачки, ведь попытки доказать родство птиц и летучих мышей не возымели успеха.
Он начал сразу с нескольких метагеномных секвенирований: что-то принадлежало кишечной микрофлоре (для интереса был взят еще и желудок коровы), что-то было с глубины почти две тысячи метров неподалеку от Калифорнии и что-то из почвенных сборов разных уголков Камчатки. Хотелось изучить как можно больше данных, ведь глобальные выводы всегда интереснее локальных!
Он сразу приступил к делу и начал смотреть на последовательности генов, сначала изучая их глазами, а потом и при помощи программ. Для более детального анализа на результаты геномных секвенирований он наложил еще и транскриптомы — все отсеквенированные молекулы матричной РНК из соответствующих организмов, они позволяют судить о том, какие гены в данный момент экспрессируются. Правда, довольно быстро было решено добавить еще и протеом — совокупные последовательности всех имеющихся белков в организме, ведь интересно, какой белок получится из той или иной РНК, если получится вообще.
При анализе всех этих «-омов» тривиальное правило подтвердилось, но со множеством исключений.
1. Со старт-кодонами всё оказалось довольно однозначно: у всех прокариот проблемы были одни и те же. С единственно возможным AUG крайне активно конкурировали по меньшей мере три варианта кодонов, которыми в 10–40% случаев начинались гены. Какие это могли быть кодоны и с чем связана такая вариабельность? Как рибосома «понимает», с какого из них начинать трансляцию, а с какого нет?
2. Первыми в истории о стоп-кодонах интерес вызвали бактерии: у целого спектра из них в геноме было не три стоп-кодона, а два: не хватало UGA. Конечно, юный биоинформатик в каждом таком случае сразу бросался искать пропажу по всему геному и сразу же находил: в рамке считывания, посреди генов, бесчисленное множество раз. С чем это могло быть связано? Как в этом случае меняются функции стоп-кодона?
3. Увиденное в конце концов натолкнуло на мысль: может ли у эукариот стоп-кодон встречаться в середине гена в норме? И ответ оказался положительным! В первом же геноме человека было найдено по меньшей мере два десятка таких генов, которые, если верить транскриптомным данным, транскрибируются в белки полноценной длины несмотря на стоп-кодон в середине. Какую роль стоп-кодон выполняет в гене, если находится в его середине? И как рибосома отличает его от настоящего стоп-кодона?
4. Кульминацией в истории со стоп-кодонами стала человеческая митохондрия: в ней не было найдено ни одного стоп-кодона, все гены заканчивались кодонами аргинина. Как могли возникнуть подобные изменения и почему именно аргинин стал выполнять функции стоп-кодона?
5. Более того, у некоторых генов функцию стоп-кодона выполнял всего один нуклеотид — тимин. Как рибосома «понимала», что именно на этом тимине нужно закончить синтез белка?
Подсказка
Бактерии без третьего стоп-кодона UGA заинтересовали нашего биоинформатика — и на его поиски в рамке считывания он отправился не только в геном, но и в транскриптом и протеом. В транскриптоме количество генов с ним несколько упало, что уже вызывало вопросы: куда пропала часть? Но и в протеоме загадок было не меньше: UGA там транслируется в триптофан: чем может быть вызвано такое изменение?
Немного поразмыслив, юный биоинформатик вернулся к задаче с митохондрией: «Между кодонами аргинина и одним из стоп-кодонов есть сходство, которое можно заметить, только если допустить, что митохондриальная рибосома может читать кодоны не только в рамке считывания, но и с небольшим пересечением», и эврика! По всей видимости, так оно и есть.
Что до старт-кодонов, то оказалось хорошей идеей посмотреть на гены не только от старт-кодона до стоп-кодона, но и за их пределами. Сразу же стало понятно, что у прокариот начало гена на матричной РНК определяется не только старт-кодоном, но и другими сигнальными последовательностями, среди которых внимание приковывала одна, находящаяся примерно на десять нуклеотидов раньше старт-кодона.
Решение
Как известно, биология отлично подтверждает известные всем слова: из любого правила всегда найдутся исключения. На этот раз они дополняют свойства генетического кода, среди которых нам интересна универсальность. Казалось бы, абсолютно у всех организмов на Земле один или несколько кодонов должны кодировать одну аминокислоту: например, триптофан может кодироваться только кодоном UGG, а известные всем UGA, UAG и UAA — это только стоп-кодоны.
Но биология не была бы биологией, если бы это всегда было так. Как мы увидели в условии задачи, порой у разных организмов один и тот же кодон (например, UGA) кодирует разное: у одних стоп-кодон, а у других триптофан. Еще чаще внутри одного организма все тот же UGA может как работать стоп-кодоном, так и кодировать другую аминокислоту — селеноцистеин.
Попробуем разобраться с этими исключениями. Посмотрим на генетический код и обратим внимание на кодоны: их сильно больше, чем аминокислот, а потому на каждую из них зачастую приходится сразу несколько кодонов. Причем иначе никак: если бы аминокислота кодировалась не тремя нуклеотидами, а двумя, всего кодонов было бы 16 — меньше, чем аминокислот. Примем имеющееся за неизбежность и попробуем найти плюсы избыточности генетического кода.
Сразу на ум приходит защита от потенциальных мутаций: если закодировать похожими кодонами одну аминокислоту, с некоторой вероятностью замена одного нуклеотида в кодоне не приведет к смене аминокислоты в белке и, как следствие, мутация ни на что не повлияет. Это уже немного снижает вероятность вредных мутаций, но на этом генетический код не остановился.
Возьмем довольно популярную в мире белков аминокислоту лейцин, она закодирована четырьмя кодонами: CUU, CUC, CUA, CUG, то есть мутация будет безвредной, только если произойдет в третьем нуклеотиде. А если в первом? Например, получится AUU или AUC — это уже совсем не лейцин! Но генетический код «предусмотрел» куда больше, ведь это крайне похожий на лейцин по структуре и тоже гидрофобный изолейцин, в белках он будет вести себя почти так же. Возьмем мутацию GUU — это такой же гидрофобный аланин. То есть избыточность генетического кода позволяет нивелировать эффект многих мутаций.
Получается, избыточность кода явно не бесполезна, и так просто менять кодоны местами не стоит. Но иногда такие перетасовки не влияют на жизнеспособность даже в случае с теми кодонами, изменения в которых будут нарушать структуру белка чаще остальных, — это стоп-кодоны. Казалось бы, их лучше сделать побольше, чтобы избавить нас от вреда многих мутаций, так как поломка стоп-кодона приведет к тому, что синтез белка с мРНК не остановится вовремя и белок выйдет длиннее, чем нормальный. Но нет, стоп-кодонов три, а это в два раза меньше, чем кодонов самой маленькой аминокислоты глицина.
Еще раз вспомним стоп-кодоны: UGA, UAG и UAA. И это не так уж и мало. В митохондриях человека стоп-кодонов еще меньше — только два, и все они другие: AGG и AGA (генетический код митохондрий сам по себе специфичен, см. Vertebrate mitochondrial code). В ядерных генах они были бы аргинином, но в митохондрии за аргинин отвечают сразу четыре кодона: CGU, CGC, CGA и CGG. Причина таких странных стоп-кодонов отлично показана для человека, сюрпризов здесь не так много: митохондриальная рибосома, по-видимому, не распознает AGG и AGA как терминирующие (см. R. Temperley et al., 2010. Hungry Codons Promote Frameshifting in Human Mitochondrial Ribosomes). Прямо перед ними ее рамка считывания сдвигается на один нуклеотид назад, а перед этими кодонами в митохондриальных генах всегда стоит U — вместе с ним эти кодоны выглядят как UAGG и UAGA, а это, если отбросить последнюю букву, стандартный стоп-кодон UAG.
Помимо этого, показано, что митохондриальные гены некоторых позвоночных (включая человека) имеют неполные стоп-кодоны, оканчивающиеся на U или UA, которые становятся стоп-кодоном UAA только при последующем полиаденилировании — достраивании к концу матричной РНК множества аденинов (см. D. Ojala et al., 1981. tRNA punctuation model of RNA processing in human mitochondria).
Но на этом сюрпризы внутри человека не заканчиваются. Порой стоп-кодоны из стандартной тройки у нас можно встретить прямо в середине гена в рамке считывания, и такие гены чаще всего относятся к двум группам. Первую называют псевдогенами — это гены, которые по тем или иным причинам перестали считываться, а потому в них можно встретить буквально что угодно, ведь на выживаемость это не повлияет. Но нас интересует вторая группа — это те гены, в середине которых стоп-кодон UGA кодирует аминокислоту селеноцистеин (отличается от цистеина атомом селена, стоящим на месте серы), таких у человека свыше двух десятков. Но на такое способны не все стоп-кодоны, а только те, за которыми следует специальная сигнальная последовательность. У нас и архей она располагается на 3'-конце мРНК и сворачивается в шпильку из примерно 60 нуклеотидов, которая и является сигналом для рибосом. У бактерий она обычно располагается сразу после кодона селеноцистеина (рис. 1).
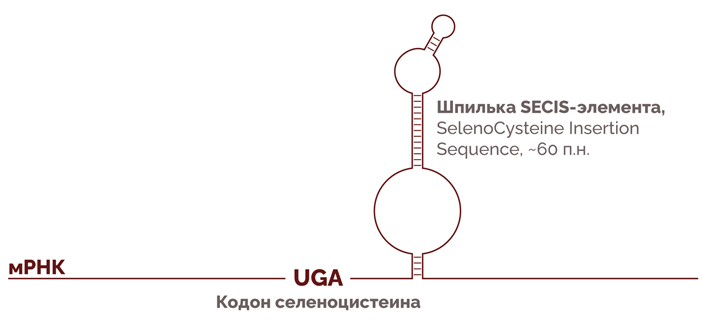
И это не единственный пример аминокислоты, которой в обычном генетическом коде нет и поэтому ей приходится делить кодон со стоп-кодоном, — к числу таких относится еще и свойственный метаногенным археям (например, Methanosarcina barkeri) и некоторым бактериям (например, Desulfitobacterium hafniense) пирролизин, который по тому же принципу кодируется уже другим стоп-кодоном, UAG.
На самом деле таких необычных случаев куда больше, чем может показаться. Возьмем двух других бактерий: практически любую из рода Mycoplasma (один из многочисленных возбудителей атипичной пневмонии) и Spiroplasma citri. Если провести простейший анализ их генов, выписывая последние три нуклеотида каждого, то в массе там будут представлены только два стоп-кодона, а вот третьего, UGA, не будет. Он дополнил набор кодонов триптофана, который обычно кодируется только похожим на него UGG. Но почему так произошло? Для рода Mycoplasma показано, что в их геномах пары A = T массово заменялись на G ≡ С, что сказалось и на стоп-кодонах (T. H. Jukes, 1985. A change in the genetic code in Mycoplasma capricolum).
Но это не единственный пример, когда UGA начинает кодировать аминокислоту, ведь у целого набора прокариот и эукариот он кодирует глицин: например, у Gracilibacteria, Hexamita inflata (эукариот из бывшей супергруппы «Excavata», паразитическая дипломонада), Streblomastix strix (родственник дипломонады — оксимонада, помогающая термитам переваривать древесину) и у некоторых зеленых водорослей (такие есть среди трибуксиевых, дазикладовых, трентеполиевых и кладофоровых).
Итого, отличия в числе стоп-кодонов мы смогли найти как у эукариот, причем самых разных, так и у бактерий и архей, но всё же одну крупную группу оставили без внимания.
Бактерии, которые сделали UGA кодирующим ту или иную аминокислоту кодоном, по сути приобрели иммунитет к некоторым вирусам, ведь теперь белки тех из них, у кого UGA стоп-кодон, не смогут синтезироваться нормально. Но и на исключение найдется исключение: предполагается, что специфичные к этим бактериям фаги тоже адаптировались и «отредактировали» генетический код на манер хозяина (N. N. Ivanova et al., 2014. Stop codon reassignments in the wild).
Может показаться, что такое происходит только вокруг стоп-кодонов, так что разберем и случаи по другую сторону гена — со старт-кодонами, которым был посвящен первый вопрос задачи. Казалось бы, AUG (метионин) — единственный вариант старт-кодона. Если посмотреть на частоту всех трехнуклеотидных сочетаний в начале каждого из кодирующих белки генов прокариот, то преобладать и правда будет AUG (в среднем около 60–70%). Но немалую долю оттянут на себя и другие, например CUG (обычно около 10%, но может доходить и до нескольких десятков процентов) и не такие многочисленные UUG и GUG (не более 10% каждый). Они отличаются лишь одной заменой в первом нуклеотиде, однако бывают и другие варианты: например, еще более редкие AUU, AUA и ACG. Как правило, все эти кодоны, если стоят в начале гена, кодируют именно метионин, а вот в любой другой части этого же гена уже свои аминокислоты. Но как же рибосома отличает два этих случая, то есть начало гена от остальных его частей?
У прокариот ответ довольно прост: по шестинуклеотидной последовательности Шайна — Дальгарно, которая стоит неподалеку от старт-кодона. С этой последовательностью при посадке связывается рибосома: на ее рибосомальной 16S РНК находится комплиментарная последовательности Шайна — Дальгарно последовательность анти-Шайна — Дальгарно (рис. 2). Если связывание пройдет правильно, то в сайте начала трансляции рибосомы окажется именно первый кодон гена — старт-кодон. Эта последовательность довольно вариабельна и бывает как «сильной», так и «слабой», и она наравне со старт-кодоном подсказывает рибосоме, где надо начинать трансляцию. И если она «сильная», то старт-кодон может отличаться от стандартного довольно сильно. А если «слабая», то есть накопила довольно много мутаций и сильно отличается от стандартной, то старт-кодон должен быть именно AUG.
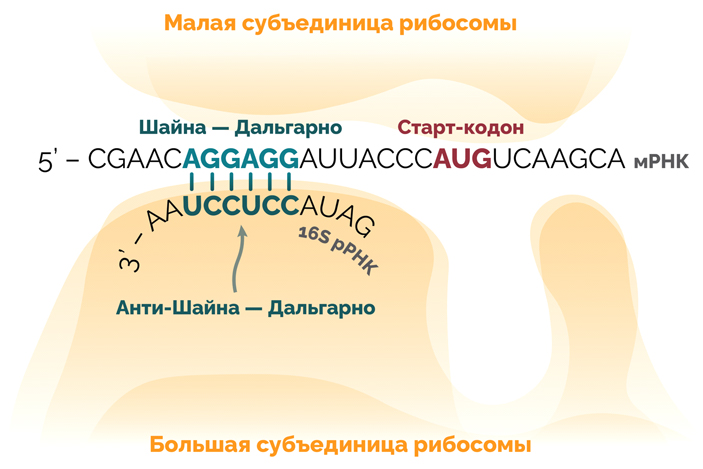
Рис. 2. Структура связывания матричной РНК с субъединицами рибосомы перед началом трансляции
У эукариот ситуация несколько сложнее и поэтому ближе к правилам: последовательности Шайна — Дальгарно нет, так что наиболее частая и практически единственная возможная замена AUG — это крайне похожий на него CUG, однако экспериментально были получены и другие работающие варианты старт-кодонов.
Но всё это лишь вершина айсберга, ведь в малых последовательностях с открытыми рамками считывания (small open reading frames, smORFs) в качестве старт-кодона могут встречаться и многие другие варианты. Ранее эти последовательности считали исключительно некодирующими, но сейчас показано, что среди них есть крупные классы важных регуляторных белков.
Послесловие
Рассмотренные исключения из стандартного генетического кода довольно четко говорят нам об одном: гены не так просты, как может показаться. Они эволюционируют так же, как и всё остальное в биологии, а с ними эволюционирует и генетический код и рибосомы.
Из-за этого найти четкие границы генов методами компьютерной биологии порой совсем не так просто: даже в обычной человеческой митохондрии самые очевидные из этих методов не сработали бы, пытаясь закончить ген одним из стандартных стоп-кодонов.
Тот факт, что методы поиска конкретных генов в геномах довольно часто так или иначе ориентируются на старт- и стоп-кодоны, до недавнего времени казался нормальным, и лишь в последнее десятилетие было показано, что из-за этого мы упускаем из вида множество важных регуляторных последовательностей: те же smORFs, которые дополнительно усложняют собственную идентификацию длиной не более чем в 100 аминокислот. При этом в геноме Drosophila melanogaster таких последовательностей тысячи, они относятся к самым разным функциональным классам, и все они ранее не включались в протеомы.
Автор благодарит С. М. Глаголева за помощь в редактировании текста задачи.
-
-
Добрый день!
Он есть в абзацах про митохондриальные стоп-кодоны, процитировал ниже.
«Помимо этого, показано, что митохондриальные гены некоторых позвоночных (включая человека) имеют неполные стоп-кодоны, оканчивающиеся на U или UA, которые становятся стоп-кодоном UAA только при последующем полиаденилировании — достраивании к концу матричной РНК множества аденинов (см. D. Ojala et al., 1981. tRNA punctuation model of RNA processing in human mitochondria)»
-
Последние задачи















Рис. 1. Вторичная структура мРНК в окрестности кодона селеноцистеина. SECIS-последовательность в 60 нуклеотидов образует сигнальную шпильку, которая и «указывает» рибосоме на то, что UGA — кодон селеноцистеина, а не стоп-кодон