Последние выпуски
- Том 86, № 5. Сентябрь-октябрь 2025
- Том 86, № 4. Июль-август 2025
- Том 86, № 3. Май-июнь 2025
- Том 86, № 2. Май-июнь 2025
«Хвост» хлоропластов отметает гипотезу об универсальности бактериальной структуры геномов

В работе сибирских биоинформатиков проведен анализ текста ДНК хлоропластов и цианобактерий методом, ранее использованным для исследования бактериальной ДНК. Метод основан на выявлении структуры текста генома по матрице часот встречаемости триплетов, составляющих геном. Другими авторами ранее было показано наличие семикластерной структуры геномов бактерий – шесть из них содержат триплеты из кодирующих последовательностей, а седьмой из некодирующей. Вопреки ожиданиям, структура геномов хлоропластов имеет значительные отличия от таковой у бактерий, например, структура генома не семи-, а восьмикластерная, нет зависимости структуры генома от GC-состава. Такие отличия структуры геномов хлоропластов, по-видимому, объясняются значительно большим по отношению к бактериальному геному количеством участков, кодирующих 16S и 23S рРНК (хлоропласты имеют 5 % от длины ДНК, бактерии - 0,5 %). Применяемый метод не выявил структурных кластеров у цианобактерий, что кардинально отличает их геном от такового хлоропластов и бактерий.
Изучение внутреннего порядка последовательностей нуклеотидов в ДНК является важнейшей задачей. Для такого рода исследований применяются различные методы и соответственно применяемым методам в молекулах ДНК выделяют разные структуры. Относительно недавно для исследования структуры геномов был предложен метод статистического анализа распределения триплетов, составляющих геном. Суть этого метода в том, что геном рассматривается как матрица частот триплетов (троек подряд идущих нуклеотидов с шагом в три нуклеотида – как будто каждый фрагмент считается транслируемым геном), составляющих полную последовательность исследуемого генома. ДНК разбивается на большое число таких формально выделяемых фрагментов и составляется матрица частот встречаемости триплетов по конкретному геному. Затем полученные матрицы анализируют методом главных компонент. Таким методом были проанализированы геномы бактерий и выяснилось, что в пространстве векторов триплеты группируются в семь кластеров (рис. 1). Эти кластеры соответствуют кодирующим фрагментам генома (шесть кластеров) и некодирующим (один кластер). Шесть кластеров возникает из-за сдвига рамки считывания (т.е. каждая буква представлена в трех триплетах, согласно поступательному движению рамки считывания, следовательно попадает в три кластера) и записи генов в ДНК в двух одноцепочечных комплементарных нитях ДНК (стрэндах), т.е. происходит удвоение троек кластеров.
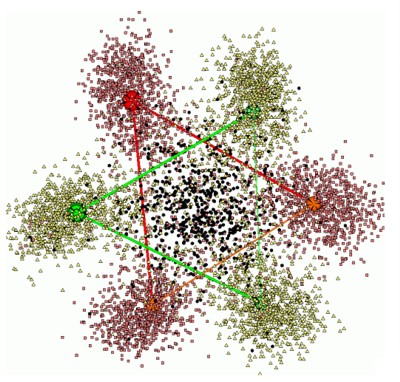
Хлоропласты рассматриваются как потомки древних бактерий (цианобактерий), перешедших к симбиотическому существованию в организмах-хозяевах; тем самым, можно ожидать, что они будут также обладать аналогичной структурой генома. Целью данного исследования стала проверка утверждения об универсальности бактериальной семикластерной структуры генома. Для выявления структуры геномы хлоропластов и цианобактерий разбивались на (пересекающиеся) интервалы длиной 603 нуклеотида, которые покрывали исходный геном с шагом в ∆ = 11 нуклеотидов. В каждом таком фрагменте подсчитывался частотный словарь триплетов, согласно описанному выше методу. Для выявления особенностей распределения определялись главные компоненты и кластеры, в которые эти точки (фрагменты) группируются.
Важнейшим результатом является выявленное отличие структуры геномов хлоропластов от аналогичных структур, выявляемых в бактериальных геномах. В отличие от семикластерной структуры геномов бактерий, у хлоропластов обособляются восемь структурных кластеров. Оказалось, что единый у бактерий кластер (седьмой), содержащий триплеты из некодирующих последовательностей, у хлоропластов имеет четкое подразделение на два отдельных кластера (заглавная иллюстрация). Дополнительный восьмой кластер, названный авторами «хвостом», содержит триплеты из фрагментов последовательностей, кодирующих 16S и 23S рРНК (РНК, входящие в состав субъединиц собственных рибосом хлоропластов), а также фрагменты, содержащие последовательности, кодирующие тРНК (транспортные РНК). Другой важной особенностью структуры геномов хлоропластов является независимость структуры генома от GC-состава. Взаимное расположение кластеров бактериальных геномов двух комплементарных одноцепочечных нитей ДНК определяется количеством GC-пар: тройки кластеров поворачиваются друг относительно друга в разных плоскостях до почти полного совмещения. В отличие от бактерий, GC-контент генома никак не влияет на детали структуры геномов хлоропластов. По-видимому, это можно объяснить значительной неоднородностью распределения GC-нуклеотидов в геноме хлоропластов, а именно, их концентрацией в «хвосте», т.е во фрагментах, несущих участки ДНК, кодирующих рРНК. Последних у хлоропластов на порядок больше, чем у бактерий (5% и 0.5% от общей длины генома, соответственно). Третьим важным отличительным моментом является то, что кластеры у геномов хлоропластов, соответствующие противонаправленным стрэндам (одноцепочечным комплиментарным нитям ДНК), всегда проектируются друг на друга и в этой проекции фрагменты, соответствующие кодирующим областям в ведущем стрэнде (нити ДНК) и имеющие сдвиг рамки считывания, равный 2, совпадают с аналогичными фрагментами из вспомогательного стрэнда; две других фазы при этом не совпадают. На рисунке (см. заглавную иллюстрацию) совпадающие фазы показаны жёлтым и оранжевым цветом (вид «анфас» и «профиль» распределения).
Анализ генома цианобактерий не выявил наличие обособленных кластеров, что отличает их как от бактерий, так и от хлоропластов.
Смотрите по теме симбиогенеза на страницах ЖОБ:
Возникновение и эволюция пластид
С 2018 полные тексты статей «Журнала общей биологии» за последнее десятилетие доступны авторизованным пользователям на ресурсе Научной электронной библиотеки eLIBRARY.RU
Популярные синопсисы





