Генетический алфавит расширен. Или нет?
Дмитрий Жарков
«Наука из первых рук» №1(61), 2015
В десятку наиболее выдающихся результатов 2014 г. вошли работы исследователей из Скриппсовского института (Ла-Холья, США), которые не только создали ДНК с двумя новыми «буквами» генетического кода, но и заставили ее воспроизводиться внутри кишечной палочки — традиционного экспериментального объекта молекулярных биологов.
Как всем известно со школьной скамьи, «текст» молекулы ДНК — хранилища наследственной информации — написан всего четырьмя «буквами»: A, T, G и C. Буквы эти обозначают четыре азотистых основания — аденин, тимин, гуанин и цитозин, которые крепятся к сахарофосфатному остову. Две такие конструкции и составляют знаменитую двойную спираль ДНК, которая в итоге содержит все инструкции по постройке и работе нашего организма.
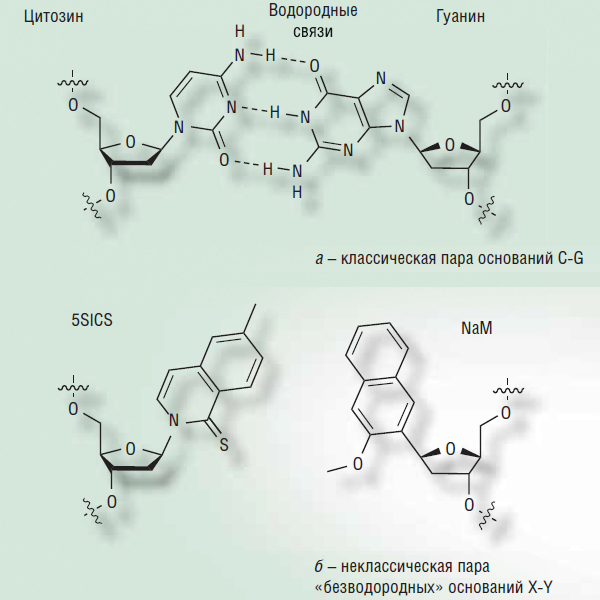
Две цепи ДНК связаны друг с другом по принципу комплементарности: напротив A в одной цепи всегда стоит T в другой, а напротив G — всегда C. Между «противоположными» основаниями в этих парах образуются так называемые водородные связи, что и определяет соответствие оснований друг другу. Например, между A и C такие связи образоваться не могут, поэтому и говорят, что основания подходят друг к другу как ключ к замку.
Структура ДНК стала известна еще в середине прошлого века, и уже тогда ученые стали задаваться вопросами: почему живая природа использует именно эти две пары оснований, а не какие-нибудь другие, и нельзя ли как-нибудь «подкрутить винтики» в клетке, чтобы заставить ее использовать другие основания? Ответ на первый вопрос не получен до сих пор. Многие специалисты в области предбиологической эволюции предполагают, что такой выбор был случайностью, которая закрепилась в дальнейшем, когда был пройден самый трудный этап возникновения жизни на нашей планете — когда молекулы стали «репликаторами», т. е. научились воспроизводить сами себя.
Вероятность такого события очень мала, поэтому неудивительно, что если даже некогда и существовали комплементарные пары с другими основаниями, они просто не прошли через это «бутылочное горлышко» эволюции. Впрочем, первой молекулой-репликатором, скорее всего, была не ДНК, а РНК, которая, кстати сказать, вместо тимина использует еще одно азотистое основание — урацил, также образующий пары с аденином. При переходе к «миру ДНК» урацил заменился на тимин по причинам, связанным с надежностью хранения информации.
Самое интересное, что и сейчас известны организмы, у которых «великолепная четверка» отличается от описанной в школьном учебнике. Например, у многих бактериофагов — вирусов, поражающих бактерии, место тимина в ДНК занимает урацил либо гидроксиметилурацил или другие производные урацила с дополнительно присоединенным углеводным остатком. Такая «подмена» помогает вирусу защищаться от охранных систем бактерий, расщепляющих проникшую внутрь чужеродную ДНК. А в 1970-х гг. в обыкновенной луже ленинградские микробиологи обнаружили бактериофаг, у которого аденин полностью заменен другим основанием, 2,6-диаминопурином.
Что касается второго вопроса, то он лег в основу нового направления молекулярной биологии — создания искусственного генетического кода. Специалисты в этой области занимаются не только поиском возможностей создания альтернативных пар оснований, но и способов введения в структуру белков неканонических аминокислот (как известно, генетические системы всех известных на сегодня живых организмов кодируют ровно 20 «стандартных» аминокислот). Понятно, что если научиться собирать ДНК из расширенного репертуара пар оснований и заложить в код возможность включения в белки нестандартных аминокислот, то это откроет невиданные перспективы перед синтетической биологией — областью науки, занимающейся созданием несуществующих в природе живых систем и процессов.
На фоне такой глобальной проблемы достижение, отмеченное журналом Science, не выглядит чем-то сногсшибательным. Скорее это — очередная ступенька лестницы, строительство которой началось два десятилетия назад, причем ступенька не очень высокая. Главный концептуальный прорыв на этом пути был совершен еще в конце 1990-х гг. группой под руководством Э. Кула (Рочестерский университет, США), которая показала, что для создания стабильной пары оснований, хорошо укладывающейся в двойную спираль ДНК, вовсе не нужны водородные связи. Можно сделать искусственные основания, вообще не содержащие ни одного атома, способного образовывать такие связи, и они смогут не только стабильно существовать в ДНК, но и без проблем включаться в нее обычными ферментами ДНК-полимеразами, по крайней мере, некоторыми из них.
В лаборатории Ф. Роумсберга, удостоенной внимания редакторов Science, неклассическими основаниями занимаются уже не первый год. Но до недавних пор все исследования в этом направлении выполнялись in vitro, т. е. «в пробирке», а не в живой клетке. В этот раз исследователи взяли одну из таких «безводородных» пар оснований и попытались заставить ее воспроизводиться внутри бактерии кишечной палочки, традиционно используемой для экспериментов молекулярными биологами.

Американским исследователям впервые удалось заставить воспроизводиться в бактериальной клетке фрагменты «химерной» ДНК, в которую были введены неканонические «буквы» — основания, названные X и Y. В качестве модели использовалась кишечная палочка E. coli. На фото вверху — скульптурное изображение кишечной палочки. Стекло. Худ. Л. Джеррам
Однако в живом организме основания не возникают по желанию экспериментатора. За каждой из четырех букв в ДНК стоит многоходовая схема их синтеза в клетке и, разумеется, основания, придуманные химиками, клетка сама делать не может. Поэтому ученые схитрили: они ввели в бактерии белок из клеточной стенки диатомовой водоросли Phaeodactylum tricornutum, который способен захватывать те самые отдельные «буквы» ДНК непосредственно из внешней среды. Соответственно, ненатуральные основания (точнее, не сами основания, а дезоксинуклеозидтрифосфаты — «кирпичики» с частью сахарофосфатного остова, из которых и строится ДНК) просто добавляли в культуральную среду, в которой росли такие бактерии.
Но существовала еще одна проблема, которую требовалось разрешить. Дело в том, что если заставить бактериальные клетки использовать ненатуральные основания в большом количестве, то бактерии просто не выживут, потому что существующий генетический аппарат не будет их «узнавать». Поэтому ученые ввели лишь одну-единственную ненатуральную пару, и то не напрямую в саму бактериальную ДНК, а в плазмиду — отдельную маленькую кольцевую молекулу ДНК, способную существовать и самовоспроизводиться внутри бактериальной клетки. А поскольку фермент ДНК-полимераза III, которая отвечает у бактерий за репликацию основной части генома, нестандартные основания вообще не узнает, ненатуральную пару оснований пришлось ставить даже не просто в плазмиду, а в очень маленький участок плазмиды, который синтезируется другим ферментом — ДНК-полимеразой I.
После всех этих манипуляций бактерии растили на обогащенной нестандартными основаниями среде в течение 15 часов — за это время клетки успевали поделиться 24 раза. Затем определяли, что находится в том месте плазмиды, где стояла ненатуральная пара. Если бы клетка не была способна при репликации использовать соответствующие друг другу неканонические нуклеотиды, а встраивала напротив них нормальные, то ненатуральная пара после 24 делений сохранилась бы только в 1 случае из 17 млн! Однако на самом деле ничего подобного не произошло: ненатуральная пара сохранялась в 86% случаев, замена произошла только спустя нескольких суток дальнейшего роста.
Важность работы Роумсберга и его коллег неоспорима, ведь им действительно впервые удалось показать работоспособность ненатуральной пары оснований в живой клетке. Но говорить о том, что в ней удалось «расширить генетический алфавит», очень и очень преждевременно — эта фраза вынесена в заголовок статьи в Nature явно с рекламными целями. В конце концов, авторы статьи обошли самые главные неразрешенные проблемы искусственного генетического кода. Ведь для того, чтобы на деле расширить алфавит ДНК, нужно как минимум встроить в клетку пути синтеза неканонических нуклеотидов, сделать их совместимыми с основной системой репликации и, главное, придумать, как при помощи новых букв заставить клетку производить и новые белки.
Задача по-прежнему выглядит чрезвычайно сложной — примерно, как полет в космос в эпоху начала аэронавтики. В этом смысле работу Роумсберга и его коллег можно сравнить с запуском шара братьев Монгольфье. Но в космос в конце концов полетели не воздушные шары, так что хотя отмеченное Science достижение — безусловно, шаг в нужном направлении, пока непонятно, приведет ли к цели именно эта дорога.
Литература:
1. Власов В. В., Воробьев П. Е. Мир РНК: вчера и сегодня // Наука из первых рук. 2012. № 3(45). С. 40–49.
2. Malyshev D. A., Dhami K., Lavergne T. et al. A semi-synthetic organism with an expanded genetic alphabet // Nature. 2014. V. 509. № 7500. P. 385–388.
-
-
стандартных аминокислот всего 20, но все триплеты уже расписаны. То есть, мы не можем просто взять и переопределить, что ААА будет кодировать не лизин, а какой-нибудь гиперлизин, потому что при этом сломаются все белки, для которых лизин закодирован как ААА. А с новыми буквами можно писать новые слова, не боясь поломать старые
-
Сдаётся мне, проще было бы «научить» рибосомы транслировать AAA в гиперлизин. А все имеющиеся в хромосомах кодоны AAA заменить на AAG.
«Учить» рибосомы придётся всё равно – и в случае применения нестандартных оснований тоже. Только придётся ещё и заставить клетку реплицировать эти основания без ошибок. А это представляется нешуточной задачей.
Автор выразил некоторый скепсис справедливо. Можно сказать и резче: исследование явно имеет целью сорвать аплодисменты, а не приблизиться к синтезу белков с нестандартными аминокислотами.-
Согласен, множество нерешенных проблем вроде т.н. "мусорной днк" просто вопиет о необходимости решения, а люди занимаются чисто популизмом. Как будто непонятно что пар-букв можно найти гораздо больше чем есть сейчас. Перенастроить всю систему клеточного синтеза даже не понимая зачем? Явно сначала нужно научится работать с имеющимся инструментарием, чем делать новый.
-
>>Сдаётся мне, проще было бы «научить» рибосомы транслировать AAA в гиперлизин.
да рибосомам-то, как я понимаю, как раз все равно, какие АК склеивать. 20+ они склеивают в одном и том же активном центре, примут и еще одну, разве что совсем экзотическая она будет.
тРНК тоже, наверное, нетрудно сделать подходящую. В общем, с трансляцией все попроще. А вот системы транскрипции и процессинга уже, может, и посложнее будет настраивать.
____________
>>А все имеющиеся в хромосомах кодоны AAA заменить на AAG.
Так теряем избыточность кода, соответственно надежность системы уменьшается. Ну и такая замена у прокариот может и возможна, а вдруг авторы в прицеле эукариот держат? Поди перелопать весь геном какой-нибудь капусты :)
___________
>>исследование явно имеет целью сорвать аплодисменты
сорвать грантов, скорей всего
-
-
-
Чисто практический выход:
Можно смоделировать клетку или даже целый фантастический организм, который будет жить только в присутствии этого не-натурального фактора.
Таким образом будет исключаться возможность их аварийного распространения. Даже если этого фактора вывалить в природу десяток миллионов тонн - всё равно через какую-нибудь пару веков все големы вымрут из-за его нехватки.
















Две цепи нашей ДНК, состоящие из сахарофосфатного остова с присоединенными к нему азотистыми основаниями, связаны друг с другом по принципу комплементарности: напротив аденина в одной цепи всегда стоит тиамин в другой, а напротив гуанина — всегда цитозин (а). Пары оснований удерживаются вместе так называемыми водородными связями. Однако стабильные пары оснований в ДНК могут формироваться и без участия таких связей (б). По: Malyshev et al., 2014